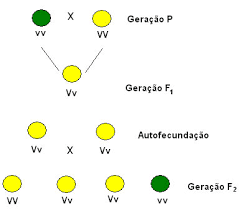
PADRÕES
CLÁSSICOS DE HERANÇA
As observações que outra eram
feitas com plantas, passaram para estudos com a Drosophila melanogaster que
impulsionou os pesquisadores a tentarem relacionar tais fatores às
características da espécie humana. Contudo a primeira barreira foi a
impossibilidade de realizar cruzamentos em laboratório.
Então começaram a analisar
gerações através dos heredogramas
para ver se os padrões de transmissão seguiam os mesmos que Mendel analisou.
Heredogramas
Podem ser tidos como diagramas
que mostram relações entre familiares,facilitando assim o estudo de determinada
característica.
Fatores
que afetam o padrão dos heredogramas
·
Heterogeneidade: Onde há diversos fenótipos que são semelhantes
mas são determinados por genótipos diferentes.
·
Penetrância: É considerado como a probabilidade de
um gene ter qualquer expressão fenotípica. Podendo ser completa e todos os indivíduos que tiverem o gene manifestarão a
característica e pode ser incompleta,
onde, mesmo possuindo o gene, nem todos manifestam a doença. Quando tratamos de
penetrância completa podemos citar como exemplo a acondroplasia (nanismo) e no
caso da incompleta se pode citar o retinoblastoma que apenas 20% das pessoas,
que possuem o gene, não manifestam a doença.
·
Pleiotropia: Assim como um fenótipo pode
influenciar vários genes, um gene pode influenciar vários fenótipos. Quando um
gene realiza esta ação, de influenciar muitos aspectos do fenótipo, ele é
chamado de pleiotrópico.
·
Expressividade: Esse termo é usado para determinar a
manifestação variada de um determinado caráter entre pessoas de um mesmo grupo
com o gene para tal característica.
HERANÇA MONOGÊNICA OU MENDELIANA
Herança autossômica dominante
Esse tipo de herança se apresenta em
múltiplas ou sucessivas gerações, afetando e sendo transmitidas tanto por
homens, quando por mulheres. É característica de mais de metade dos padrões da
herança mendeliana.
 Um exemplo desse tipo de herança é a
pseudoacondroplasia (um tipo de nanismo) onde as pessoas com a altura normal
são genotipicamente d/d e aqueles que
manifestam possuem genótipo D/d ou D/D.
Um exemplo desse tipo de herança é a
pseudoacondroplasia (um tipo de nanismo) onde as pessoas com a altura normal
são genotipicamente d/d e aqueles que
manifestam possuem genótipo D/d ou D/D.
Nesse podemos ainda observar a dominância completa, ou seja, o
indivíduo heterozigoto apresenta o caráter condicionado por um alelo dominante
e o alelo recessivo só se expressa em homozigose e a dominância incompleta, onde o indivíduo heterozigoto vai apresentar
um padrão diferente dos homozigotos e surge um fenótipo intermediário. No
último caso, teremos uma proporção fenotípica de 1:2:1 e uma proporção
genotípica de 1:3:3:1 (seguindo a
lei de Mendel).
Há ainda a sobredominância, onde o heterozigoto expressa um fenótipo mais
produtivo e superior, sendo melhor para a indústria.
Herança
dominante ligada ao X
Nesse tipo de herança os homens
afetados transmitem a condição para todas as filhas, mas não para os filhos. E
as mulheres heterozigotas afetadas casadas com homens não afetados transmitem a
condição para metade de seus filhos e filhas. E há ainda os alelos letais que,
quando em homozigose, levam o individuo à morte.
Herança
autossômica recessiva
Diferente
da herança autossômica dominante, essa ocorre em uma única geração e pode haver
irmãos onde em apenas um é expresso o caráter. Ocorrendo em homozigose ou em
heterozigose composta, que são pessoas que contém alelos diferentes para a
mesma deficiência.
Herança
recessiva ligada ao X
Quando a recessiva é ligada ao X nenhum dos filhos de homens
afetados apresentará a deficiências. Contudo, filhas serão “portadoras”,
carregando o alelo recessivo e então poderá transmitir para seus descendentes
homens através do cruzamento XAXa
×
XAY. Nesse
caso, uma mulher só terá o fenótipo se o pai e a mãe possuírem o gene (p. ex., XAXa
× XaY).
Herança ligada ao X
Podemos
usar para analise desse tipo de herança a coloração dos olhos de Drosophilas melanogaster.
 Foram realizados dois
experimentos, o primeiro fora iniciado com o cruzamento de machos com olhos
brancos e fêmeas com olhos vermelhos, o que originou toda a prole F1 com olhos vermelhos. Ao realizar o
cruzamento entre F1, se pôde observar que todas as fêmeas possuíam olhos
vermelhos e apenas 50% dos machos possuíam olhos brancos e gerando a proporção
fenotípica de 3:1.
Foram realizados dois
experimentos, o primeiro fora iniciado com o cruzamento de machos com olhos
brancos e fêmeas com olhos vermelhos, o que originou toda a prole F1 com olhos vermelhos. Ao realizar o
cruzamento entre F1, se pôde observar que todas as fêmeas possuíam olhos
vermelhos e apenas 50% dos machos possuíam olhos brancos e gerando a proporção
fenotípica de 3:1.
No segundo experimento cruzaram-se
fêmeas de olhos brancos(Xw X Xw) com
machos de olhos vermelhos(Xw+ X
Y) e a F1 observada foi que todos os
machos foram de olhos brancos e todas as fêmeas de olhos vermelhos. Ao realizar
o cruzamento entre F1 a proporção fora diferente do primeiro experimento.
Obteve-se 50% de fêmeas e machos com olhos brancos e 50% de fêmeas e machos com
olhos vermelhos.
Assim fora possível definir que o
gene que determinava a coloração branca w,
estava em dose dupla nos cromossomos sexuais das fêmeas, a ponto de
transmitir um deles para os machos. E no momento do cruzamento entre F1, ainda
na formação dos gametas, ambos doam o cromossomo sexual com a característica e
no cruzamento observamos a proporção.
PADRÕES DE HERANÇA NÃO CLÁSSICAS
Herança pseudo-autossômica
É o padrão observado em genes que estão presentes na região pseudo autossômica
dos cromossomos X e Y que podem realizar crossing-over durante a gametogênese
masculinas e passar suas característica para a prole masculina.
MOSAICISMO
Pode ser entendido como a presença em um indivíduo ou em um tecido de
células de duas linhagens diferentes geneticamente, mas que foram derivadas de
um único. Possuindo células com marcação paterna e células com marcação
materna. Ainda durante o processo de divisão pré ou pós-natal que dará origem
ao organismo mosaico, podem surgir mutações que podem estar presentes em alguns
tecidos do corpo, mas não nos gametas e serem chamadas de mosaicismo somático puro, podem estar apenas nos gametas e em
nenhum outro lugar e serem chamados de mosaicismo de linhagem germinativa puro
ou estar presente tanto em células somáticas quato em células germinativas. O
que derteminará onde estará esse mosaicismo será o momento que ocorre a
mutação, se ocorrer antes do momento de segregação das células, todas a células
terão mosaico para a mutação; caso contrário, o local será determinado
dependendo do local para onde a célula se segregou.
O mosaicismo somático ocorre quando a mutação, ainda no período
embrionário, ocorre em células que darão origem a células somáticas, já o mosaicismo da linhagem germinativa se
originou de células que dariam origem em células germinativas. Ele afeta apenas a
linhagem germinativa do indivíduo, de maneira que o indivíduo não será afetado
pelo distúrbio relacionado com a mutação, mas poderá passá-la para os
descendentes, onde estará presente em todas as células.
Imprinting genômico
O imprinting genômico (também conhecido como imprinting parental) é um processo
epigenético normal onde alguns genes se expressam em apenas um alelo, enquanto
o outro é inativado (metilado). Consiste em uma forma reversível de ativação de
um gene que segue um determinado padrão (parterno ou materno) durante a
formação dos gâmetas. São conhecidas algumas doenças relacionadas com o imprinting genômico, como a Síndrome de Prader-Willi e Síndrome de Angelman.
Dissomia uniparental
Ocorre quando uma pessoa recebe duas cópias de uma cromossomo do mesmo
parental. Pode ser isodissomia quando
receber duas cópias dos homólogos e pode ser heterodissomia quando vier
uma cópia de cada homólogo.
HETERODISSOMIA
ISODISSOMIA
Mutações dinâmicas
 As mutações dinâmicas consistem
na expansão do número de unidades repetitivas, tipicamente constituídas por
tripletos, presentes num determinado gene ou na sua vizinhança.
As mutações dinâmicas consistem
na expansão do número de unidades repetitivas, tipicamente constituídas por
tripletos, presentes num determinado gene ou na sua vizinhança.
Em condições normais, um
indivíduo é portador de um número reduzido de tripletos repetidos
sequencialmente. Quando o número de repetições ultrapassa um limiar crítico a
mutação torna-se patogénica, sendo esta a causa de várias doenças genéticas. Na
expansão de diferentes tripletos o limiar crítico é diferente.
A severidade da doença e a idade a que os
primeiros sintomas surgem depende do número de repetições do tripleto.
A antecipação é um fenómeno associado às mutações dinâmicas. Consiste
no aumento de gravidade da expressão de um determinado gene numa família, em
gerações sucessivas. A idade de manifestação da doença pode também ser
antecipada ao longo das gerações.
A pré-mutação
é um estádio intermédio entre o normal e a expressão fenotípica da mutação em
que o número de repetições é além do limite normal, mas ainda abaixo do limiar
crítico.
Como este número de repetições é
muito próximo do limiar crítico, a probabilidade da descendência vir a
manifestar a doença aumenta devido ao fenómeno da antecipação.
O deslizamento na replicação do DNA
e o crossing over entre repetições desalinhadas são duas fontes possíveis de
expansão.
Este tipo de mutação pode
conduzir a perda de função ou com ganho de função tóxica da proteína (s).
Expansões associados com perda de
função são tipicamente maiores do que as expansões que correspondem a um ganho
de função tóxica da proteína.
A repetição de diferentes
tripletos leva a diferentes patologias.